Localización subcelular
Para determinar la localización subcelular in vivo de las proteínas RA1 y similares a RA1 del maíz y S. viridis , generamos una fusión traduccional N-terminal con GFP. Las construcciones 35S:GFP:RA1 y similares a 35S:GFP:RA1 se analizaron mediante un sistema de expresión transitoria en hojas jóvenes de tabaco agroinfiltradas. La fluorescencia se observó a través de un microscopio confocal de barrido láser. Se utilizó tinción DAPI para visualizar la localización de los núcleos. Como se muestra en , las proteínas RA1 y similares a RA1 se localizan en los núcleos de las células epidérmicas del tabaco.
Figura 3. Análisis de la localización subcelular de las proteínas RA1 y similares a RA1. Localización subcelular de las construcciones RA1-GFP, SvRA1-GFP, SvRA1-like A-GFP y SvRA1-like B-GFP en células epidérmicas de hojas de tabaco. El color verde es la señal de la proteína GFP. El color azul representa el núcleo teñido con DAPI.
Análisis de elementos reguladores que actúan en cis entre las secuencias promotoras RA1 y similares a RA1
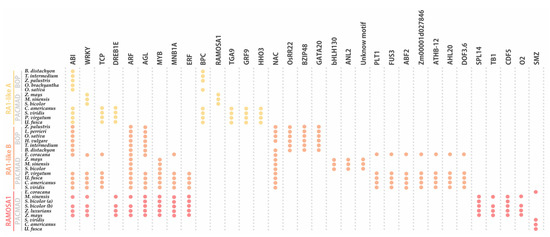
Cuando se examinaron los promotores de los genes RA1 y similares a RA1 de 16 especies de gramíneas, se identificaron muchos motivos reguladores y se agruparon en 33 tipos de elementos, de acuerdo con la familia de factores de transcripción (TF) correspondiente ( , Tabla S5 ). Se reconocieron secuencias conservadas que contenían motivos de unión para reguladores transcripcionales bien conocidos. Identificamos motivos TF involucrados en diferentes procesos biológicos relacionados con (1) la germinación de semillas, como el desarrollo del embrión, la embriogénesis somática y la regulación positiva de la proliferación de la población celular; (2) el desarrollo de la planta, como la regulación de la formación de brotes secundarios, el desarrollo de las hojas, el desarrollo de las flores y el desarrollo de las raíces; (3) la respuesta al estrés abiótico, como la respuesta al estrés salino, la respuesta al estímulo luminoso y la respuesta al agua; y (4) la respuesta al estrés biótico, como la respuesta de defensa a las bacterias ( Tabla S6 ). Además, identificamos sitios de unión de TF relacionados con la regulación positiva o negativa de las vías de señalización hormonal, los procesos biosintéticos de hormonas, así como las respuestas de auxina, giberelina, ácido abscísico y etileno ( Tabla S6 ).
Figura 6. Elementos cis no codificantes identificados en las regiones promotoras previstas de los genes RA1 y similares a RA1 de especies de gramíneas. Los círculos de colores representan la presencia de motivos conservados en el promotor. Las secuencias de consenso de motivos se muestran en la Tabla S5 .
Los motivos reguladores conservados albergaban nueve sitios putativos de unión de TF para el linaje A similar a RA1, 22 sitios putativos de unión de TF para el linaje B similar a RA1 y 13 sitios putativos de unión de TF para el linaje RA1. Encontramos dos motivos conservados compartidos por las secuencias promotoras similares a RA1 y RA1 : (1) los motivos ABI están presentes entre al menos 24 de las 35 secuencias promotoras analizadas; (2) los motivos WRKY se identificaron en 12 de las 35 secuencias promotoras.
RA1-like A comparte (1) motivos TCP con las secuencias promotoras de RA1-like B del clado PACMAD, excepto las de Andropogoneae, y (2) motivos DREB1E con las secuencias promotoras de Andropogoneae de RA1 .
RA1-like B tiene cinco motivos en común con RA1 : (1) los motivos ARF están presentes en todas las secuencias promotoras RA1-like B y RA1 de la tribu Andropogoneae; (2) los motivos AGL están bien conservados en las secuencias promotoras RA1-like B , excepto en la tribu Andropogoneae, mientras que los motivos AGL están restringidos a la tribu Andropogoneae en el linaje RA1; (3) los motivos MYB se identificaron en las secuencias RA1-like B de las secuencias promotoras PACMAD y RA1 de la tribu Andropogoneae; (4–5) el motivo MNB1A y los motivos ERF se identificaron entre las secuencias promotoras RA1-like B del clado PACMAD, excepto las de Andropogoneae, y las secuencias promotoras RA1 de la tribu Andropogoneae.
Descubrimos que cada copia del linaje de especies se caracteriza por elementos cis únicos . El linaje A similar a RA1 ha conservado motivos BPC fuera de la tribu Andropogoneae y un motivo de unión a RA1 en la tribu Andropogoneae. El motivo TGA9, el motivo GRP9 y el motivo HHO3 se identificaron solo en secuencias promotoras de especies del clado PACMAD fuera de la tribu Andropogoneae. El linaje B similar a RA1 se caracteriza por la presencia de motivos NAC en todas las secuencias promotoras analizadas, excepto en la de T. intermedium . El motivo OsRR22, el motivo BZIP48 y el motivo GATA20 están restringidos al clado BOP. Entre las especies del clado PACMAD, se identificaron los motivos bHLH130, ANL2 y un motivo desconocido en la tribu Andropogoneae, mientras que los motivos PLT1, FUS3, ABF2, Zm00001d027846, ATHB-12, AHL20 y DOF3.6 se identificaron fuera de la tribu Andropogoneae. Finalmente, las secuencias promotoras del linaje RA1 se caracterizan por la presencia de secuencias (1) motivos SPL14, TB1, CDF5 y O2 en la tribu Andropogoneae y (2) motivos SMZ fuera de la tribu Andropogoneae.