- Noticias / Un mejor serodiagnóstico de infecciones por Trypanosoma vivax en bovinos revela altas tasas de infección en las regiones ganaderas de Argentina
PLOS Neglected Tropical Diseases
Un mejor serodiagnóstico de infecciones por Trypanosoma vivax en bovinos revela altas tasas de infección en las regiones ganaderas de Argentina
Artículo
Compartir en
redes sociales
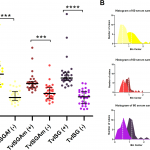
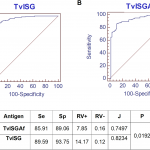
La tripanosomosis bovina, causada por Trypanosoma vivax , afecta actualmente al ganado y tiene un impacto económico significativo en África subsahariana y Sudamérica. El desarrollo de nuevos antígenos de diagnóstico es esencial para mejorar y refinar los métodos existentes. Nuestro estudio evaluó la eficacia de dos antígenos recombinantes en la detección de anticuerpos específicos en el ganado. Estos antígenos son derivados de una glicoproteína de superficie invariante (ISG) de T . vivax . Se evaluó una fracción de un antígeno previamente descrito (TvY486_0045500), designado TvISGAf, de una cepa africana, y se identificó un nuevo antígeno ISG de un aislado americano, TvISGAm. Los dos antígenos se expresaron como proteínas de fusión en Escherichia coli : TvISGAf se fusionó a la etiqueta MBP-His, y TvISGAm se obtuvo como una proteína fusionada a la etiqueta His. Se realizó una evaluación ELISA utilizando estos antígenos en 149 muestras bovinas positivas y 63 negativas. El rendimiento diagnóstico fue mejorado por el uso de una combinación de ambos antígenos (denominado ELISA basado en TvISG), logrando una sensibilidad del 89,6% y una especificidad del 93,8%. Luego de la validación del ELISA basado en TvISG, se determinó la seroprevalencia de la infección por T . vivax en 892 muestras de campo de bovinos en la región central de Argentina. La seroprevalencia media de T . vivax fue del 53%, con una variación que osciló entre el 21% y el 69% entre los seis departamentos estudiados. Estos resultados respaldan el uso del ELISA TvISG como una valiosa herramienta serológica para la detección y el monitoreo de la infección por T . vivax en bovinos. Además, informamos por primera vez la seroprevalencia de T . vivax en Argentina, lo que resalta la naturaleza endémica generalizada de la enfermedad en la región. Para controlar eficazmente la creciente propagación de T . vivax en las vastas áreas de producción ganadera de América del Sur, es esencial implementar programas de vigilancia consistentes y adoptar estrategias preventivas.
Resultados
Análisis in silico de la secuencia que codifica una glicoproteína de superficie invariante de T. vivax y su expresión recombinante
La información disponible en la base de datos del proyecto genoma T. vivax Y486 ( http://tritrypdb.org/tritrypdb/ ) contiene un marco de lectura abierto de 1203 pb (TvY486_0045500) que codifica una glicoproteína de superficie invariante de 400 aminoácidos con una masa molecular teórica de 44,5 kDa, S1 Fig ., analizada previamente por Fleming et al., 2016 [ 24 ]. Con base en esta información, buscamos en los datos genómicos de un aislado americano de T. vivax [ 29 ] un gen putativo que codifica una glicoproteína de superficie invariante. Utilizando la herramienta BLASTn y la secuencia de nucleótidos de TvY486_0045500, identificamos una secuencia de nucleótidos que codifica una glicoproteína de superficie invariante putativa (tig00000163, [ 29 ]). La secuencia de aminoácidos se presenta en S2 Fig . El análisis in silico predijo que las glicoproteínas de superficie invariantes de T. vivax africanas y americanas tienen una estructura de dominio ISG típica con un péptido señal N-terminal, un dominio transmembrana y un dominio intracelular ( Figs. S1 y S2). El análisis de la secuencia de aminoácidos de las ISG de T. vivax africanas y americanas reveló que estas proteínas comparten ~63% de identidad y ~69% de similitud ( Fig. S3 ).
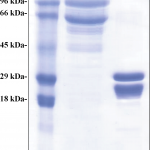
Con base en información previa [ 24 ] y predicciones de epítopos lineales ( Fig. S4 ), amplificamos secuencias ISG truncadas de ADN genómico de T. vivax africano (TvISGAf) y americano (TvISGAm) por PCR (sin el péptido señal y el dominio transmembrana N-terminal) . Las secuencias de ADN resultantes se clonaron en el vector pGEM-T Easy para su análisis. Para caracterizar la funcionalidad antigénica de las proteínas recombinantes, primero intentamos expresar los antígenos recombinantes (TvISGAf, del aminoácido 126 al 400 y TvISGAm, del aminoácido 125 al 274) en E. coli como proteínas de fusión His-tag o N-terminal MBP His-tag usando los plásmidos pET28 y pMOMAL, respectivamente. Después de evaluar las diferentes condiciones de inducción, sólo pudimos obtener el antígeno TvISGAm como una proteína de fusión con doble His-tag N- y C-terminal y el TvISGAf como una proteína de fusión con un His-tag MBP N-terminal. Estos sistemas de expresión nos permitieron obtener con éxito la sobreexpresión de los antígenos recombinantes en forma soluble. Por lo tanto, continuamos las evaluaciones serológicas utilizando los antígenos recombinantes obtenidos por estas dos estrategias. Dificultades similares con la expresión recombinante del antígeno derivado de la cepa africana se han descrito previamente [ 24 ]. Las secuencias de aminoácidos de las proteínas recombinantes se muestran en la Fig. S5 . El análisis SDS-PAGE mostró que las proteínas recombinantes se produjeron y aislaron con alta pureza electroforética ( Fig. 2 ). Sin embargo, se observó una banda de menor masa molecular aparente con abundancia relativa similar en ambos casos ( Fig. 2 ). La co-purificación de un producto truncado podría explicar este resultado, ya que las etiquetas de purificación se ubican hacia el extremo N-terminal de las proteínas recombinantes. Las proteínas recombinantes purificadas se almacenaron a -80 °C durante al menos 8 meses.